Líneas de investigación:
En el Laboratorio de Ingeniería Enzimática y Nanobiotecnología (LIENbio) combinamos técnicas experimentales y teóricas de Ingeniería de Proteínas, Biofísica y Bioinformática para el desarrollo de herramientas moleculares con aplicación Bionanotecnológica y el estudio de procesos de interés Biológico. Dentro de los temas en desarrollo actual se encuentra el mejoramiento de enzimas con actividad lignocelulolítica para la producción de biocombustibles, el desarrollo de nanopartículas multifuncionales, el estudio comparativo de la dinámica estructural de proteínas de organismos extremófilos, el desarrollo de antígenos para el diagnóstico, tratamiento y prevención de SARS-COV2, y más.
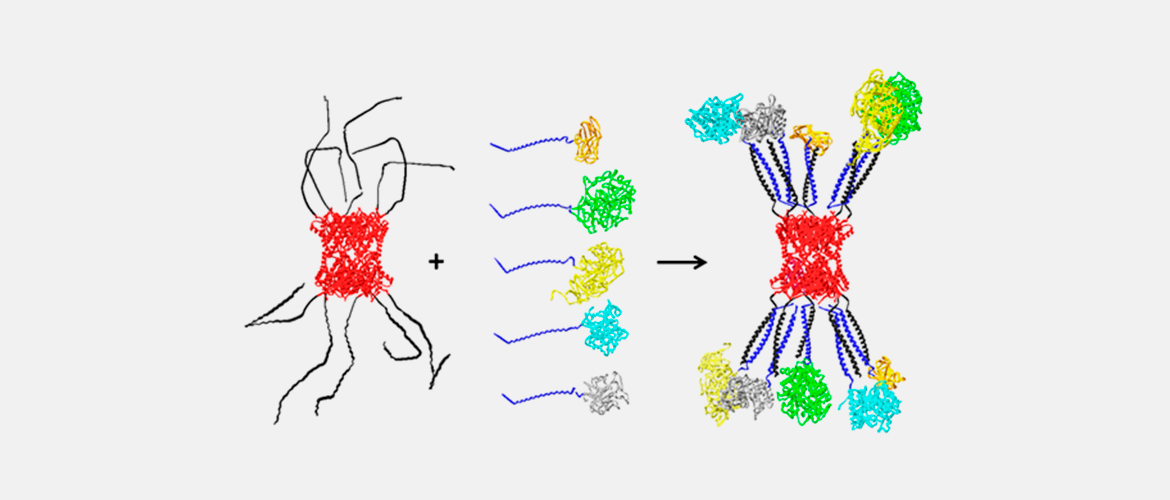
Desarrollo de complejos multienzimáticos para el mejoramiento de la degradación de lignocelulosa. Aplicación a la producción de biocombustibles
Los celulosomas producidos por ciertos microorganismos representan la maquinaria natural más eficiente para la degradación de la compleja matriz de lignocelulosa. Esto se debe al sinergismo producido por el agrupamiento espacial de diversas enzimas y dominios de unión al sustrato. Estos complejos multienzimáticos constituyen una maquinaria potencialmente útil para la producción de bioetanol. Sin embargo, la producción biológica en escala industrial de celulosomas naturales presenta serias limitaciones. Nuestro objetivo es el desarrollo y optimización de celulosomas artificiales basados en la utilización de la estructura decamérica de la proteína Lumazina Sintasa como plataforma para el agrupamiento de dominios de unión a celulosa y diversas enzimas con actividad celulasa y hemicelulasa. Es esperable que estos complejos multienzimáticos permitan aumentar la actividad de las enzimas utilizadas, reduciendo el costo de producción de bioetanol y favoreciendo la sustitución de combustibles fósiles.
Análisis de la degradación multienzimática de celulosa mediante modelos de reacción difusión
Los “modelos basados en agentes”, los “autómatas celulares” y los modelos de “reacción difusión ” son métodos de simulación numéricos en donde partículas difusibles dentro de un espacio tridimensional interactúan entre sí a través de reglas probabilísticas simples y programables según su proximidad espacial y colisiones estocásticas. La evolución temporal de estos sistemas da lugar a propiedades emergentes complejas, difíciles de anticipar de otra manera. Desarrollamos modelos de reacción difusión para la simulación de la degradación multienzimática de celulosa. Modelamos sustratos celulósicos y partículas con actividad endoglucanasa, exoglucanasa, beta glucosidasa y de unión al sustrato. Utilizamos esta herramienta para interpretar resultados experimentales de degradación de celulosa por celulosomas artificiales, y predecir los parámetros cinéticos y estructurales que son críticos para el mejoramiento de su actividad.
Diseño y producción de mejoras en la actividad y estabilidad de enzimas.
La estabilización estructural y funcional de enzimas usadas en procesos industriales es un tema de mucho interés aplicado. Esta permite aumentar la vida media de las enzimas en biorreactores o la temperatura de procesos con el consiguiente aumento de la velocidad de reacción y la mejora de costos de producción.
La combinación de métodos de ingeniería de proteínas para aumentar la estabilidad y/o actividad específica de enzimas individuales, y la producción de complejos multienzimáticos como el propuesto en otros proyectos de nuestro laboratorio, constituyen estrategias complementarias para el desarrollo de potentes celulosomas artificiales que podrían superar incluso la actividad de los celulosomas naturales. Utilizamos herramientas experimentales y bioinformáticas para el diseño de mejoras en la actividad y/o estabilidad de enzimas mediante su mutagénesis. Abordamos el diseño racional de mutantes y la evolución dirigida de enzimas claves en la degradación de celulosa y hemicelulosa, con especial énfasis en enzimas con actividad xilanasa. Además del valor aplicado de esta tarea, sus resultados constituyen una fuente de información muy valiosa para interpretar la relación existente entre la estabilidad estructural, flexibilidad conformacional y actividad de enzimas.
Estudio de la dinámica estructural de proteínas de organismos extremófilos
El estudio de proteínas homólogas de organismos extremófilos representa un excelente sistema para evaluar la dinámica funcional de las proteínas, su dependencia con la temperatura y las características de las adaptaciones evolutivas asociadas. Usamos herramientas bioinformáticas para evaluar de qué manera la estabilidad y flexibilidad conformacional de variantes extremófilas se relaciona con su plegabilidad y frustración energética. Nuestro enfoque abarca el uso de potenciales estadísticos de interacción entre residuos para el análisis de la energía de estructuras nativas y no nativas de las variantes estudiadas, y simulaciones de dinámica molecular con modelos de grano grueso para evaluar propiedades de su paisaje energético de plegamiento. Dentro de los modelos estudiados se encuentra la proteína adenilato quinasa de organismos psicrófilos, mesófilos y termófilos del género Bacillus.
Estudiantes motivados/as, becarios doctorales y postdoctorales interesados/as contactarse con el Dr. Patricio Craig.
Structural and functional comparison of SARS-CoV-2-spike receptor binding domain produced in Pichia pastoris and mammalian cells. Sci Rep. 2020 Dec 11;10(1):21779. doi: 10.1038/s41598-020-78711-6.
https://www.nature.com/articles/s41598-020-78711-6
Maidana SS, Craig PO, Craig MI, Ludwig L, Mauroy A, Thiry E, Romera SA.
Evidence of natural interspecific recombinant viruses between bovine alphaherpesviruses 1 and 5. Virus Res. 2017 Oct 15;242:122-130.
https://www.sciencedirect.com/science/article/abs/pii/S0168170217302976?via%3Dihub
Boffi JC, Marcovich I, Gill-Thind JK, Corradi J, Collins T, Lipovsek MM, Moglie M, Plazas PV, Craig PO, Millar NS, Bouzat C, Elgoyhen AB. Differential Contribution of Subunit Interfaces to α9α10 Nicotinic Acetylcholine Receptor Function. Mol Pharmacol. 2017 Mar;91(3):250-262. doi: 10.1124/mol.116.107482.
https://molpharm.aspetjournals.org/content/91/3/250
Dalamón VK, Buonfiglio P, Larralde M, Craig P, Lotersztein V, Choate K, Pallares N, Diamante V, Elgoyhen AB. Connexin 26 (GJB2) mutation in an Argentinean patient with keratitis-ichthyosis-deafness (KID) syndrome: a case report. BMC Med Genet. 2016 May 4;17(1):37. doi: 10.1186/s12881-016-0298-y.
https://bmcmedgenet.biomedcentral.com/articles/10.1186/s12881-016-0298-y
* PICT-2017-3925 – ANPCyT “Complejos multienzimáticos para el mejoramiento de la degradación de lignocelulosa y la producción de biocombustibles”.
* UBACYT 2018 “Programando el plegado de proteínas repetitivas”.